近日,中国科学技术大学瞿昆教授课题组开发了一种基于深度矩阵分解的空间表观组数据去噪算法SPEED。该算法能够以高质量的图谱级单细胞表观数据作为先验,准确实现空间表观数据去噪并辅助一系列下游分析,如表观空间区域识别、基因活性分析等。研究成果以《Denoising spatial epigenomic data via deep matrix factorization》为题,发表于2026年1月13日的《自然-计算科学》(Nature Computational Science)杂志。
近年来空间表观组学技术快速发展,出现了空间ATAC-seq、空间CUT&Tag等多种技术方法,能够在保持细胞空间位置信息的情况下对基因组表观遗传状态进行高通量测量。这些技术已广泛应用于小鼠胚胎、大脑组织以及人类样本等多种生物体系中,为理解组织内的基因调控机制提供了新的视角。然而,目前的空间表观数据普遍存在信号检测水平偏低、噪声高以及峰矩阵极度稀疏等问题。这些问题限制了空间表观技术在复杂疾病组织等情景中的应用。因此,迫切需要开发高效的数据降噪和分析方法,以提高空间表观组学数据质量并充分挖掘其中的生物学信息。
目前主流的表观组学数据分析方法在处理空间表观组数据时存在明显局限,无法有效利用空间组织结构和外部参照数据来提升建模准确性。瞿昆教授课题组提出了基于深度矩阵分解的SPEED算法,通过引入空间位置信息和单细胞表观组图谱数据先验,有效降低原始空间表观组学数据中的噪声干扰。SPEED算法自动学习大型单细胞参考图谱中基因组峰与峰之间的内在关系,并将这种知识迁移到空间数据中,从而有效缓解极度稀疏和高噪声带来的影响。与此同时,SPEED利用空间上邻近取样点的分布规律,并可选地融合对应组织切片图像的特征信息,实现对数据的多模态增强,进而保持原始数据中的空间连续模式和结构。得益于这种深度矩阵分解模型结合先验的策略,SPEED在降噪和特征提取方面优于传统无监督方法,能够产出更清晰可靠的表观组学信号。
研究人员将SPEED应用于四组模拟空间表观组学数据以及十四个真实组织切片涵盖spatial-ATAC-seq、spatial-Mux-seq、MISAR-seq、spatial-CUT&Tag等多种空间表观组实验技术。SPEED在数据降噪、染色质可及性差异分析、表观空间域识别以及基因活性分析等多个任务上表现出色,明显优于其他同类算法。
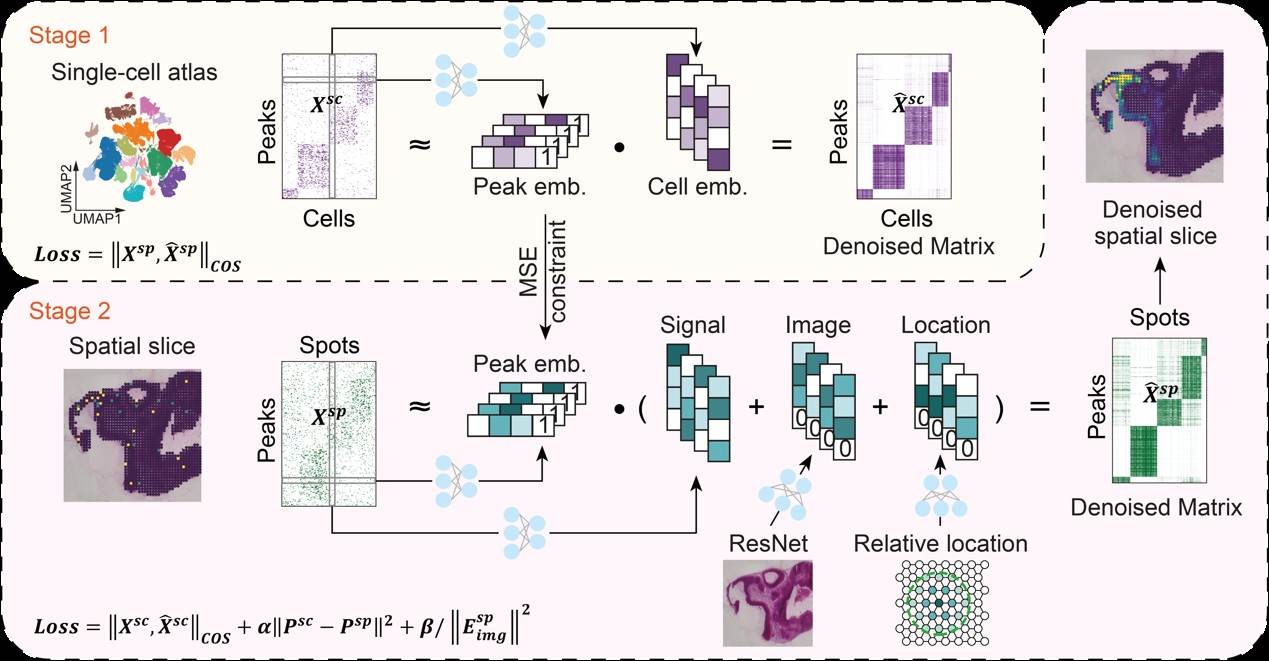
图1. SPEED算法的工作流程
瞿昆教授为本文的通讯作者,人工智能与数据科学学院博士生王姝妍和苏州高等研究院博士后许浩为本文的共同第一作者。这一研究工作得到了基金委杰出青年基金、基金委青年学生基础研究项目(博士生)、科技部国家重点研发计划、基金委自然科学基金以及中国科学技术大学原创探索类项目等多个项目的资助。
论文链接: https://www.nature.com/articles/s43588-025-00941-3
(生物医学工程学院、人工智能与数据科学学院 )
